mRNA 생산
SARS-CoV-2의 출현 이후, 전세계 과학계에서는 이 바이러스를 연구하기 위해 압도적인 노력을 기울이고 있으며, 현재 다양한 치료 및 예방을 위한 연구가 진행되고 있다. 이 중 백신 개발에서는 기존의 전통적인 형태의 백신 외에 새로운 기반의 백신들이 연구되고 있다. 백신과 같은 생명과학 제품의 제조에는 세세하게는 수백개의 복잡한 공정 단계가 필요하며 최적화된 제조법을 개발하는데 상당한 시간과 비용이 소모된다. 따라서 백신을 개발하는 생명과학 회사는 상품 비용을 줄이고, 허가 기간을 단축하고, 질병 발생에 더 빨리 대응하여 자본 효율성을 제고할 수 있는 새로운 생산 방법을 원하게 된다. mRNA 기반 백신은 이러한 백신의 제조 과제를 해결하고 새로운 조성물을 제공할 수 있을 것으로 기대되는 대표적인 형태라 할 수 있다. 중심원리(Central dogma)의 중간 유전물질 인 mRNA를 기반으로 한 약물의 개념은 1989년 R. W. Malone에 의해 여러 형태의 진핵 세포에 mRNA가 성공적으로 transfection 되어 발현될 수 있다는 것이 알려지면서 개발 가능성이 재조명되기 시작했다. mRNA 백신은 기존 백신이 갖지 못한 많은 장점을 가지고 있는데, 생산 및 제조 측면에서는 다음과 같은 장점이 있다. 우선, mRNA는 이론적으로 모든 종류의 단백질을 암호화할 수 있다. 이는 mRNA 염기 서열을 변형하여 백신 개발 효율을 최적화할 수 있다는 것을 의미하는데, 이는 다른 형태의 백신에 비해 편리한 방법이다. 여기에서 중요한 부분은, 대부분의 mRNA 백신 생산 및 정제 과정은 서로 다른 항원을 사용하더라도 매우 유사하기 때문에 다른 종류의 mRNA 백신을 개발하기 위해 설비가 유지되거나 표준화되어 사용될 가능성이 있다는 것이다. 따라서 mRNA 백신 생산은 시간과 경제적 비용면에서 상당한 메리트를 갖고 있다.
mRNA 백신이 임상적으로 효과적이고 안전하다고 가정하면, 어떤 감염병이 발생했을 때 이에 대해 긴급히 대응할 수 있다는 이점이 존재한다. mRNA 백신은 몇 주 내에 항원을 인코딩하는 서열을 포함한 임상 배치를 생산할 수 있다. 아래에서 mRNA 제조에 대하여 살펴보자.
1. mRNA 합성: in vitro transcription과 template DNA 제작 공정
현재 가장 널리 사용되는 mRNA 합성 방법은 T3, T7 또는 SP6 RNA 중합효소와 주형(template) DNA를 사용하여 세포를 사용하지 않는 in vitro transcription을 진행하는 것이다. 따라서 mRNA 합성 공정에는 RNA 중합효소가 주형으로 삼는 DNA가 필요하다. 그러므로, 주형 DNA의 생산부터 mRNA 제작 공정이 시작된다고 볼 수 있다. In vitro transcription에 사용하는 주형 DNA에는 RNA 중합효소가 인지할 수 있는 프로모터를 포함하고 있어야 한다. 이 주형 DNA는 RNA 중합효소에 의해 전사 반응을 진행한 후 DNase를 처리하는 공정에서 제거되나, 후에 기술할 정제과정에서 제거할 수도 있다. 현재 제시되는 주형 DNA 제작 방식은 DNA를 대장균에서 증식 가능한 플라스미드 DNA를 사용하는 것과 선형 DNA 형태를 PCR로 증폭하여 사용하는 방식으로 나눌 수 있다. 대장균에서 플라스미드 DNA를 제작하는 방식은 대장균을 배양하는 기술과 플라스미드 DNA를 추출하는 공정을 필요로 하며, 이후 DNA를 선형화 하는 공정을 거치게 된다. 세포를 사용하는 배양공정 및 세포 수집, 파쇄, 플라스미드 DNA 추출 등의 공정을 수립해야 하며, mRNA 생산과 독립된 별도의 DNA 생산 공정 및 설비를 갖춰야 한다. 전체적으로 보면 단순하지 않은 공정을 가지게 되나, 대장균 배양 및 DNA 정제의 경우는 현재 상업적으로 사용 가능한 시설들이 잘 갖춰져 있으며, 규모가 큰 상업용 생산이 가능하다. 한편 주형 DNA를 PCR로 증폭하여 사용하는 방식은 선형 DNA를 in vitro 반응으로 증폭할 수 있으므로 플라스미드 DNA 제작 공정에 비해 상대적으로 간단하나, PCR 반응을 위한 DNA 중합효소, primer, dNTP 등의 추가적인 재료들을 필요로 하며, 상업화로 사용하기 위한 큰 scale의 생산이 어렵다. 또한 뒤에 기술될 poly (A) tail이 삽입된 template DNA를 생산하기 어렵다는 단점도 지니고 있다. 현재 시점에서는 상업용 스케일의 생산 공정은 대장균을 사용한 플라스미드 DNA를 사용하는 쪽으로 기울어져 있으나, 각각의 방식이 장단점을 가지고 있으므로 향후 전체적인 공정의 시간 및 비용의 편익과 합성 산물의 정확도(fidelity)에 따라 유리한 방식으로 전환이 이루어질 것으로 기대된다.
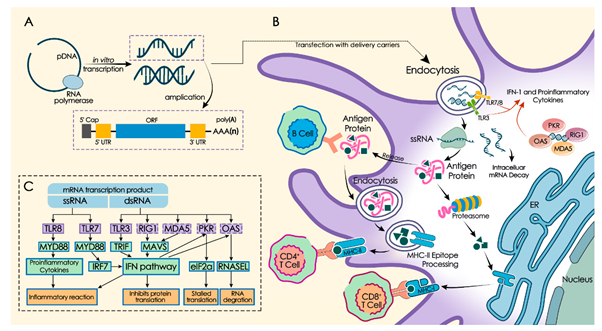
<mRNA in vitro transcription 및 innate immunity>
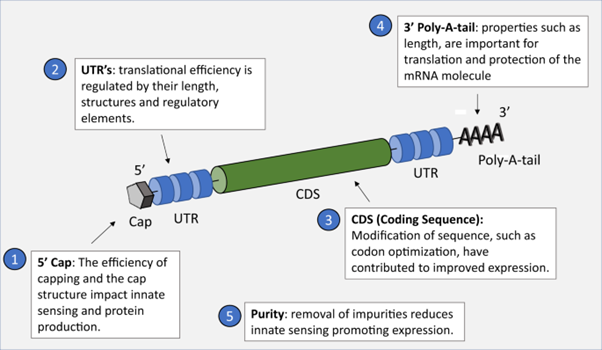
<mRNA 구조>
2. mRNA 구조 및 최적화
mRNA의 구조를 변형하는 여러 전략들을 살펴보기에 앞서 mRNA가 지니는 면역원성 특징을 일부 살펴볼 필요가 있다. mRNA가 가지는 특징 중 하나는 자체적인 면역증강효과(self-adjuvanting effect)를 포함하는 것이다. mRNA를 외래 유전자의 벡터로 사용할 때, mRNA 바이러스와 유사한 특성을 나타낼 수 있다. 이 경우 mRNA는 항원 제시 세포(APC)에 의해 인식될 수 있으며, 이는 이후 Toll-like receptor 3 (TLR3), TLR7 및 TLR8과 같은 pattern recognition receptor (PRR)를 활성화한다. In vitro transcription의 산물로 일부 포함될 수 있는 double strand RNA (dsRNA)는 RIG-I 및 MDA5와 같은 세포질의 수용체 와 결합할 수 있으며, 이후 APC의 maturation 및 type I IFN, proinflammatory cytokine의 분비를 촉진하여 항원 특이적 체액성 및 세포성 면역반응을 유도할 수 있다. Single strand RNA (ssRNA) 형태의 mRNA는 세포내에서 번역되고, 번역된 단백질은 세포 외부로 방출되어 B cell 면역반응을 유도하거나, MHC에 의해 T cell 면역반응을 유도할 수 있고, TLR7 및 TLR8 인식을 통해 dendritic cell의 항 바이러스 효과를 유발할 수 있다. 그러나, 세포질에서 mRNA에 의해 자극된 과도한 면역 반응은 세포가 대량의 type I IFN 및 기타 인터페론을 분비하도록 자극하여 mRNA의 번역을 억제하고 결국 RNA 분해, CD8+ T cell 감소를 유발할 수 있으며 이는 궁극적으로 면역 반응 감소로 이어질 수 있다. mRNA의 자가 면역증강 특성은 mRNA를 백신으로 사용하는 데에 장단점을 모두 가지고 있기 때문에 다양한 의학적 요구에 따라 mRNA 면역원성을 조절하는 것이 필요하다.
5’ capping:
mRNA 구조에는 기본적으로 5’ cap, 5’ UTR, ORF, 3' UTR 및 poly(A) tail의 구조를 지니며 온전한 구조를 갖도록 합성하는 것은 mRNA의 안정성 및 발현 효율에 영향을 준다. 진핵세포 및 이에 기생하는 바이러스의 mRNA 중 일부는 5' 말단에 7-methyl guanosine (m7G) 캡을 갖는다. 이는 5', 5'- triphosphate bridge (ppp)를 통해 첫 번째 RNA 뉴클레오티드에 연결되고 이후 전사과정이 진행된다(m7GpppGpN 구조). 5' cap은 mRNA의 안정성을 향상시켜 ribosome이 mRNA의 시작점을 인식하고 번역 개시 인자(eIF4E)에 결합하여 번역 효율을 향상시킬 수 있게 한다. mRNA capping에는 일반적인 두 가지 방식이 있다. 첫째, mRNA in vitro transcription 시에 m7G nucleotide (m7GpppG)와 같은 Cap analogue를 추가하면 transcription과 함께 mRNA capping이 진행된다. Cap analogue를 사용한 방법은 mRNA in vitro transcription의 가장 일반적인 방법이지만, Cap analogue가 mRNA 전사 시작 시에 역방향으로 결합할 수 있음이 밝혀졌다. 이 경우, Gpppm7GpN 형태의 cap을 가진 mRNA 이성질체가 형성되는데, 이러한 형태의 mRNA는 번역의 효율이 낮다. 5’ cap의 역결합을 피하기 위해 anti-reverse cap analogs (ARCA)가 개발되었다. 일반 Cap analogue에 비해 ARCA로 capping 된 mRNA는 더 높은 번역 효율을 보인다. 최근에는 mRNA의 안정성을 높이고, 번역 효율을 개선하기 위해 ARCA 구조에 대한 추가적인 연구들이 진행되고 있다. 대규모 제조의 경우 5' capping은 일반적으로 cap analogue를 사용하여 수행한다. 일반적으로 80% 정도의 효율을 보이는데, 바꿔 말하면 20% 정도의 capping 되지 않은 mRNA가 생산되므로 공정 생산성 감소 및 효능 감소로 이어질 수 있는 부담을 지니고 있다. 다른 방법으로는 in vitro transcription 후 효소 반응을 통하여 capping 할 수 있다. 백시니아 바이러스의 capping complex 등을 사용하여 반응을 진행할 수 있는데, 이 capping complex는 triphosphatase, guanylyltransferase 및 (guanine-7-)methyltransferase 활성을 지니고 있어 mRNA의 5’에 capping을 진행할 수 있다.
Untranslated region (UTR)의 최적화:
UTR은 mRNA 코딩 영역의 upstream (5' UTR) 및 downstream (3' UTR) 도메인에 위치한 mRNA 서열의 비 코딩 부분이다. UTR은 mRNA 복제 및 번역 과정과 관련이 있으며 RNA 결합 단백질을 통해 mRNA 안정성 및 번역 효율에 크게 영향을 미칠 수 있다. 일반적으로 UTR 최적화는 생체 내 mRNA 발현 수준을 높이고자 진행되는데, 3' UTR은 mRNA의 불안정한 영역으로 여겨지므로 3' UTR에 불안정한 서열을 피하면 mRNA 안정성을 높일 수 있고, 반감기를 늘릴 수 있다. 5' UTR은 downstream에 위치하는 ORF의 번역에 직접적인 영향을 미치므로 5' UTR의 최적화는 ORF의 정상적인 번역 과정에는 영향을 주지 않아야 한다. 5' UTR에 ORF의 시작부분과 유사한 유전자 서열을 피하면 mRNA 번역 시에 frame의 잘못된 시작을 효과적으로 방지할 수 있다. 또한, mRNA의 안정성과 번역의 정확성을 향상시키기 위해 일부 특정 서열을 5' UTR에 추가할 수 있다. 예를 들어 Kozak 시퀀스를 삽입하여 번역 프로세스를 보다 정확하게 시작하도록 구성할 수 있다. 한편, 일부 연구에 따르면 5' UTR이 과도하게 안정화되는 2차 구조를 형성할 경우, 리보솜이 mRNA에 결합하는 것을 방해하여 번역 효율을 떨어뜨릴 수 있다는 보고가 있으므로, 5’ UTR는 적절한 안정성과 발현 효율을 보이는 최적화 과정이 필요하다.
ORF의 최적화:
mRNA에서 단백질을 코딩하는 영역인 ORF 영역 또한 번역 효율에 상당한 영향을 미친다. 이 영역에서 적절한 codon을 선택하면 mRNA의 전체 번역 효율을 최적화할 수 있다. 일반적으로 ORF의 codon 최적화는 희귀 codon을 빈번히 사용되는 codon으로 치환하게 된다. 이 경우 세포 내에 풍부하게 존재할 것으로 예상되는 tRNA를 사용하게 되기 때문에 번역 효율이 증가할 수 있다. 그러나 일부 단백질은 정확하고 안정적인 folding을 위해 낮은 번역 속도를 필요로 하는 경우가 있다. 때문에 mRNA의 높은 번역 속도가 모든 경우에 유리한 것은 아니다. 오히려 이러한 경우에는 ORF에서 빈도가 낮은 codon을 사용하여 고품질의 단백질을 생산할 수 있다. 따라서 서로 다른 항원의 경우 mRNA 번역 속도 향상과 동시에 발현된 항원의 품질을 보장하기 위해 서로 다른 codon 최적화 전략을 사용해야 한다.
ORF 영역에서 염기의 변형은 PRR 인식에 의한 면역 반응을 감소시키고 mRNA의 번역을 향상시킬 수 있다. Pseudouridine으로 변형된 mRNA와 변형되지 않은 mRNA의 차이를 연구한 결과에 의하면 변형된 mRNA는 type I INF 유도하는 능력이 낮고, RNA 분해 효율이 낮아 안정성이 높으며 따라서 번역 효율이 더 높은 것으로 나타났다. Uracil analog는 mRNA 변형에 사용되는 가장 일반적인 유사체이며 cytidine등 일부 다른 염기 유사체도 mRNA 서열 변형에 사용될 수 있다.
poly(A) tail 구조:
poly(A) tail 서열은 RNA exo-nuclease의 분해 과정을 늦출 수 있으며, 이는 안정성을 증가시키고 생체 내 반감기를 연장하며 mRNA의 번역 효율을 향상시킬 수 있다. 또한, poly(A) binding protein (PABP)은 eIF4G 및 eIF4E와 같은 번역 개시 인자를 통해 5' cap에 연결될 수 있으며, 이는 차례로 mRNA의 loop 구조에 영향을 미치고 mRNA의 안정성 및 번역 효율을 향상시킬 수 있다. 그러나 PABP는 microRNA에 의해 매개되는 번역 억제 과정에 참여할 수 있으므로 다양한 Poly (A) tail 길이가 mRNA 번역 효율에 다르게 영향을 미칠 수 있다. Poly(A) tail 구조를 합성하는 방법에는 여러 가지가 있으며, 그 중 Poly (A) 구조 정보를 가진 DNA 템플릿을 사용한 in vitro transcription 은 정해진 길이의 Poly (A) tail을 합성할 수 있어 흔히 사용하는 방법이다. 재조합 Poly (A) 중합 효소는 mRNA 전사 후 추가되어 poly (A) tail을 추가할 수 있으며, 이 방법은 다양한 길이의 poly (A) tail이 합성될 수 있다. 초기 연구에서는 긴 Poly (A) tail이 mRNA 안정성을 향상시킬 수 있었으며, Poly (A) tail 길이가 20개 미만이면 mRNA의 번역 효율이 감소하는 결과를 보여주었다. 그러나 최근의 유전체 기반의 연구에서 높은 번역 효율을 가진 mRNA는 오히려 짧은 Poly (A) tail을 갖는다는 것을 발견하였다. 따라서 다양한 유형의 세포에서 높은 번역 효율 mRNA에 필요한 Poly (A) 염기 서열의 길이가 다를 수 있고, mRNA의 번역 효율을 최적화하기 위해서는 poly (A) tail 길이에 대한 최적화가 이루어져야 한다.
3. mRNA의 정제
mRNA의 in vitro transcription 후 정제는 일련의 접선 유동 여과(Tangential flow filtration, TFF) 단계를 통해 달성될 수 있으며, 90%에서 99.9%의 순도 및 90%~95%의 수율로 설정 가능하다. TFF는 size exclusion, ion exchange 및 poly dT 기반의 affinity 크로마토그래피 등의 정제 기술로 보완되어 구성될 수 있다. 앞서 기술된 template DNA를 제거하는 관점에서는 플라스미드 DNA는 PCR product를 사용하는 방식보다 크기 및 전하비율 등에서 크로마토그래피를 통한 제거가 용이하다. mRNA의 in vitro transcription 산물에 double strand RNA (dsRNA) 형태가 포함될 가능성은 앞서 언급하였는데, RNA 바이러스가 복제될 때의 중간체와 비슷한 dsRNA는 type I IFN 생산을 촉진할 수 있다. 따라서, in vitro transcription 생성물은 크로마토크래피를 사용한 과정을 통해 type I IFN 면역 반응을 감소시킬 수 있는 방향으로 정제되며, 정제된 mRNA는 번역 효율 또한 증가될 수 있다. 생성된 mRNA는 지질 나노 입자(lipid nano particle, LNP)로 제조된다. LNP 공정 및 제형은 RNA 서열과 무관하며, 따라서 서로 다른 용도의 mRNA 백신에 공통적으로 사용될 수 있다.
mRNA 기술의 특성은 다른 서열을 필요로 하는 백신에 대해 서열상의 거의 무한한 조합으로 빠른 개발을 가능하게 하는데 있다. 이를 위해서는 후속 개발을 위한 최적의 구조, 제형, 공정을 선택해야 한다. 예를 들어, 불활화 및 약독화 바이러스 백신이나, 재조합 단백질 백신 후보는 제품 별 제조 공정을 개발하고 최적화, 검증 및 cGMP 생산이 승인되어야 하며, 이 과정에는 상당한 시간이 걸릴 수밖에 없다. 반면 mRNA 백신은 시퀀스만 변환하면 (물론, template DNA를 생산하는 대장균 균주를 개발해야 하지만) 전체적인 공정 프로세스의 수정 또는 검증이 용이하여 상대적으로 빠른 개발이 가능하다. 또한, 단위 부피 및 단위 시간당으로 표현되는 생산성은 mRNA 플랫폼에서 기존의 시스템보다 상당히 높게 나타난다. mRNA 백신 생산 공정은 시설 규모 면에서 기존의 백신 생산 공정보다 1/2~1/3 규모로 운용 가능하며, 자본 투자 관점에서는 1/20 ~ 1/35의 초기 비용만을 요구한다. 따라서 보다 빠르고 용이하게 건설할 수 있고, 일회용 장비를 광범위하게 사용할 수 있는 소규모 시설로 가능하다는 강력한 이점을 가지고 있다. 일회용 장비를 사용하여 공정을 구축하면 이후 다른 시퀀스를 사용한 백신 생산 시에 생산 설비의 전환이 용이하므로 공정 및 품질 관리의 설계 및 개발 일정을 단축할 수 있는 장점을 갖는다. Covid-19와 같이 신속한 대응이 필요한 경우, 기존의 대규모 시설을 재배치함으로써 전통적인 형태의 백신을 생산하여 대응하는 전략을 세울 수 있으나, 수요를 충족하기 어렵거나 다른 의약품 생산에 부정적인 영향을 미치게 된다면 추가 시설의 건설이 시급히 필요하게 된다. 이러한 재래식 시설의 건설로 수 년의 시간과 수백 억 이상의 자본이 필요하다면, mRNA와 같은 신규 플랫폼 기술은 신속한 대응 및 제조에 상당한 이점을 제시할 수 있을 것이다. mRNA 플랫폼을 통해 얻을 수 있는 시간과 자원의 이득은 이 플랫폼이 완전히 개발된 미래에는 더 명백해질 것으로 예상된다.
<참고자료>
Xu, S.; Yang, K.; Li, R.; Zhang, L. mRNA Vaccine Era—Mechanisms, Drug Platform and Clinical Prospection. Int. J. Mol. Sci. 2020, 21, 6582. https://doi.org/10.3390/ijms21186582
Jackson, N.A.C., Kester, K.E., Casimiro, D. et al. The promise of mRNA vaccines: a biotech and industrial perspective. npj Vaccines 5, 11 (2020). https://doi.org/10.1038/s41541-020-0159-8
Pardi, N., Hogan, M., Porter, F. et al. mRNA vaccines — a new era in vaccinology. Nat Rev Drug Discov 17, 261–279 (2018). https://doi.org/10.1038/nrd.2017.243
Kis, Z., Kontoravdi, C., Dey, A. K., Shattock, R., & Shah, N. (2020). Rapid development and deployment of high?volume vaccines for pandemic response. Journal of Advanced Manufacturing Processes, 2, e10060. doi: 10.1002/amp2.10060.
Zhuang X, Qi Y, Wang M, Yu N, Nan F, Zhang H, Tian M, Li C, Lu H, Jin N. mRNA Vaccines Encoding the HA Protein of Influenza A H1N1 Virus Delivered by Cationic Lipid Nanoparticles Induce Protective Immune Responses in Mice. Vaccines (Basel). 2020 Mar 10;8(1):123. doi: 10.3390/vaccines8010123. PMID: 32164372; PMCID: PMC7157730.
Wu G, Huang C, Yu YT. Pseudouridine in mRNA: Incorporation, Detection, and Recoding. Methods Enzymol. 2015;560:187-217. doi: 10.1016/bs.mie.2015.03.009. Epub 2015 Apr 27. PMID: 26253972; PMCID: PMC5702494.
Stepinski J, Waddell C, Stolarski R, Darzynkiewicz E, Rhoads RE. Synthesis and properties of mRNAs containing the novel 'anti-reverse' cap analogs 7-methyl(3'-O-methyl)GpppG and 7-methyl (3'-deoxy)GpppG. RNA. 2001 Oct;7(10):1486-95. PMID: 11680853; PMCID: PMC1370192.
US 2016/0024547 A1
이전
2021.01.07
다음
2021.03.09